第一步:进入你之前创建的nnUNetFrame文件夹里面,创建一个名为DATASET的文件夹,现在你的nnUNetFrame文件夹下有两个文件夹,nnUNet是代码源,另一个DATASET就是我们接下来用来放数据的地方; 第四步:进入右边文件夹nnUNet_raw_data,创建一个名为Task08_HepaticVessel的文件夹(解释:这个Task08_HepaticVessel是nnUNet的作者参加的一个十项全能竞赛的子任务名,你可以对这个任务的数字ID进行任意的命名,比如你要分割心脏,你可以起名为Task01_Heart,比如你要分割肾脏,你可以起名为Task02_Kidney,前提是必须按照这种格式) nnUNet是如何知道你的文件存放在哪儿呢,当然要在环境中创建一个路径,按照我的步骤,别做更改,因为到现在为止,你的路径和我的路径是一致的。 第四步:在home下打开终端,输入 ② Task008的imagesTr和labelsTr内: 最少需要8g显存,一轮的时间很慢,在11g2080ti上时间一轮为550s,作者训练1000轮为一个实验结果,时间很慢,需要付出多一点耐心。 在这个框架里,调参侠是没有尊严的,你引以为傲的各种trick,在这里并没有太大作用,这就是为什么我奶奶都能用的原因。你只需要这三行命令,然后等待1000轮结束,请在训练的时候翻翻我之前的博客,毫无疑问那才是nnUNet的魅力所在。 你太懒了,这个交给你自己琢磨吧!
Follow Me, Buddy!
一、写在前面
二、nnUNet框架如何安装?
1. 你应该配置哪些环境?
pytorch和anaconda的安装应该不用多说,这里着重说一下,安装torch时装上的cuda10.1为阉割版,会不利于我们接下来安装Apex,所以请安装完整版的,B乎的这个还可以cuda10.1安装。
当我后面想玩一下TensorRT的时候,发现TensorRT7不支持cuda10.1,所以有其他需求的,装cuda10.2也ok。
这是英伟达的一个用于混合精度训练的插件,请不要直接pip,跟着下面的操作来:
第一步:打开Apex所在项目网站,往下拉便可以看到QuickStart,已经很详细。
第二步:在你用来安装环境的目录下打开终端,git clone https://github.com/NVIDIA/apex;
第三步:cd apex 进入你刚才下载下来的apex文件夹里面
第四步:pip install -v --no-cache-dir --global-option="--cpp_ext" --global-option="--cuda_ext" ./【这步出现问题尝试使用 python setup.py install --cuda_ext --cpp_ext ,更多问题参考这里】
当你在安装好cuda的情况下完成这四步时,应该不会有问题,我用阉割版的cuda就会出现安装失败的问题。如果有问题请在下方评论反馈。
pip install --upgrade git+https://github.com/nanohanno/hiddenlayer.git@bugfix/get_trace_graph#egg=hiddenlayer(没有换行,这是一行代码)
第一步:在home下创建nnUNetFrame文件夹,在这个文件夹内打开终端git clone https://github.com/MIC-DKFZ/nnUNet.git
第二步:cd nnUNet
第三步:pip install -e .(兄弟们和集美们别忘了加 . )
当你安装完成这些以后,你的每一次对nnUNet的操作,都会在命令行里以nnUNet_开头,代表着你的nnUNet开始工作的指令。
2. 整理你的数据!
① nnUNet需要你把你要训练的数据做一个好好的整理,初学者请务必按照我的做法,等你熟练掌握以后再考虑新的姿势(有些文件夹的创建时多余的,但是你还是跟着我这样做最好):
第二步:进入创建好的DATASET文件夹下面,创建下面三个文件夹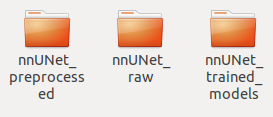
第二个用来存放原始的你要训练的数据,第一个用来存放原始数据预处理之后的数据,第三个用来存放训练的结果。
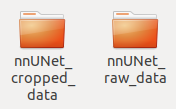
第三步:进入上面第二个文件夹nnUNet_raw,创建下面两个,右边为原始数据,左边为crop以后的数据。
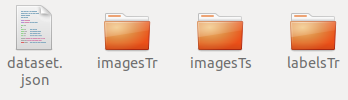
第五步:将下载好的公开数据集或者自己的数据集放在上面创建好的任务文件夹下,下面还以作者参加的Task08_HepaticVessel竞赛为例,解释下数据应该怎么存放和编辑:
3.设置nnUNet读取文件的路径
第一步:在home目录下按ctrl + h,显示隐藏文件;
第二步:找到.bashrc文件,打开(vim当然可以,但是毕竟教给我奶奶用的);
第三步:在文档末尾添加下面三行,右上角保存文件,观察下面保存成功后关闭。export nnUNet_raw_data_base="/home/qiao/nnUNetFrame/DATASET/nnUNet_raw" export nnUNet_preprocessed="/home/qiao/nnUNetFrame/DATASET/nnUNet_preprocessed" export RESULTS_FOLDER="/home/qiao/nnUNetFrame/DATASET/nnUNet_trained_models" source .bashrc来更新该文档。
nnUNet已经知道怎么读取你的文件了。
三、在Task08_HepaticVessel上进行训练!
1. 转换一下你的数据集,让它可以被nnUNet识别
nnUNet_convert_decathlon_task -i /home/qiao/nnUNetFrame/DATASET/nnUNet_raw/nnUNet_raw_data/Task08_HepaticVessel
转换之后会发现,在这个Task08_HepaticVessel文件夹旁边多了一个Task008_HepaticVessel,对的,这说明框架已经安装成功了,之后的操作要在这个文件夹上进行。
让我们看一下这两个文件夹里面的训练文件有什么不同:
① Task08的imagesTr和labelsTr内:
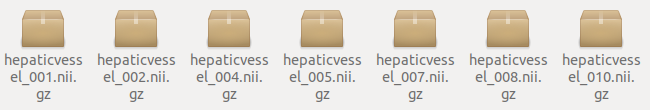
可以看出文件末尾多了_0000,是的,这就是你的数据格式是否正确的标志。不仅训练需要这个格式,之后你在推理的时候,也应当把你的文件名设置为这样,后面我会详细的说。
③ jason文件的解释:
json文件中包含着你的训练数据信息和任务信息:
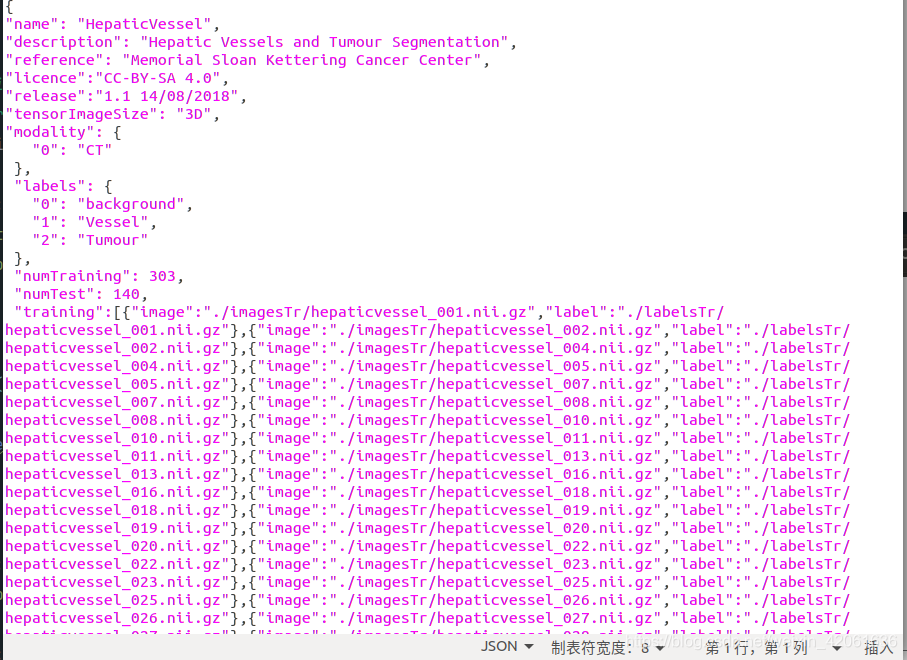
尤其注意上图中的labels,里面有0 、1 、2三个标签,这是因为Task08的任务是分割出肝血管和肝血管瘤,三分类自然有三个标签。可以看到这个任务的训练集为303个,测试集为140个(用来进行模型训练好以后的推理测试,而不是验证!)自然,如果你需要做2分类,请自行去掉2这个标签,nnUNet会自动识别你应该做什么几分类。
建议将数据集都下下来进行观察,有些的模态也不一样。2. 预处理
nnUNet_plan_and_preprocess -t 8
因为你的任务ID为8,所以参数t为8。这个过程会消耗很多的时间,速度慢的原因在于对要进行插值等各种操作。预处理要求你一定一定一定要在SSD【固态硬盘】上进行,Task08预处理后的数据会占据大约100多个g,请保证自己的SSD足够大,空间不够请自己筛选一部分的训练文件,不要200多套全拿来训练。3.开始训练
nnUNet_train 3d_fullres nnUNetTrainerV2 8 4
8代表你的任务ID,4代表五折交叉验证(0代表一折)。所有的任务都应当在“4”的情况下,也就是五折交叉验证下进行。具体的参数和作用,我会在详谈训练的新博客中进行解释。迄今为止,我共用3d_fullres + nnUNetTrainerV2的方式训练了十个分割模型,除了本身在论文中表现就较差的模型外,大多数的分割效果都较为理想,请相信你手中的工具。4.简单说下配置
5.至于调参
四、怎么在自己的训练集上训练?
本网页所有视频内容由 imoviebox边看边下-网页视频下载, iurlBox网页地址收藏管理器 下载并得到。
ImovieBox网页视频下载器 下载地址: ImovieBox网页视频下载器-最新版本下载
本文章由: imapbox邮箱云存储,邮箱网盘,ImageBox 图片批量下载器,网页图片批量下载专家,网页图片批量下载器,获取到文章图片,imoviebox网页视频批量下载器,下载视频内容,为您提供.
阅读和此文章类似的: 全球云计算
 官方软件产品操作指南 (170)
官方软件产品操作指南 (170)